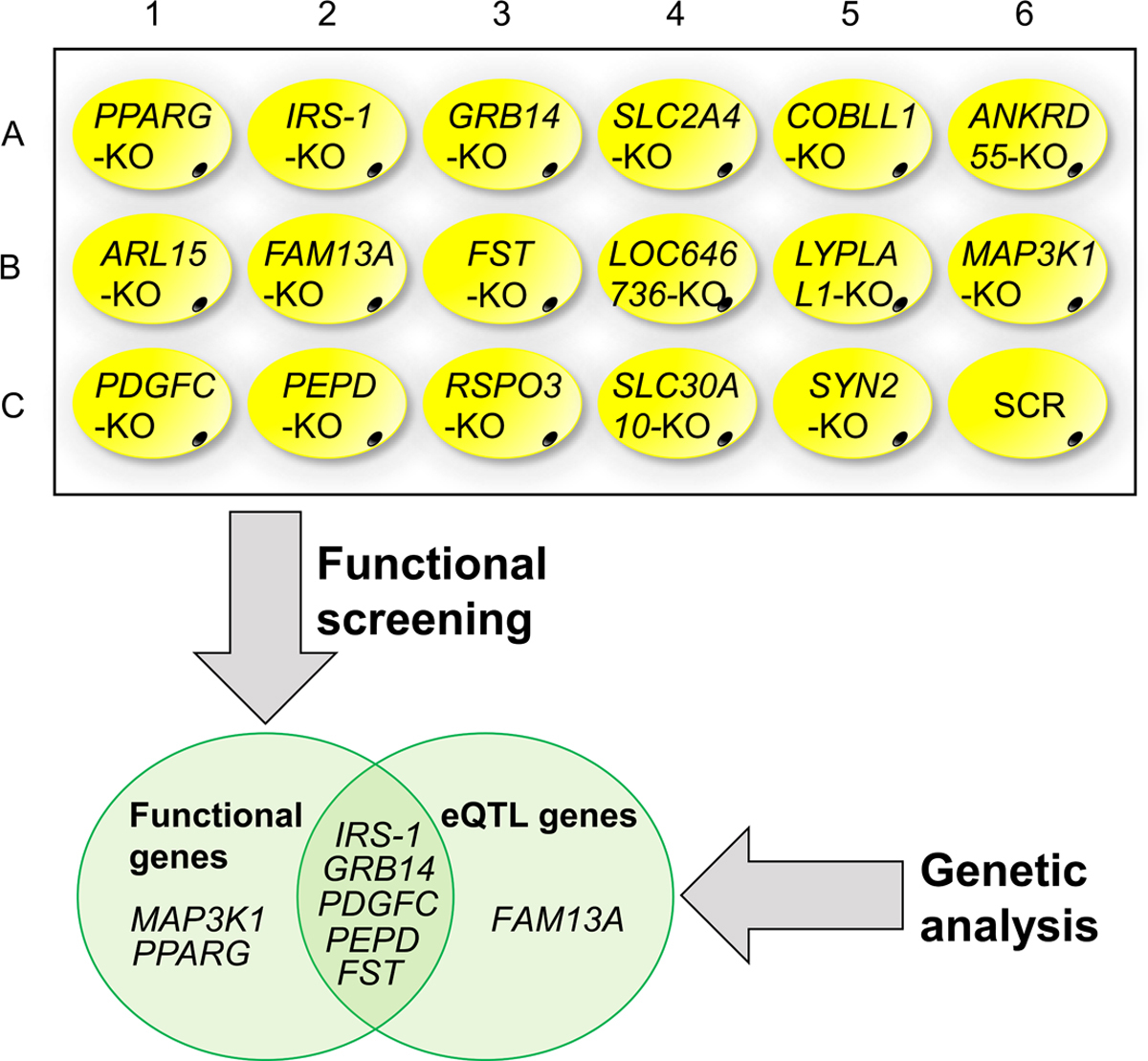
为了识别胰岛素抵抗(IR)的候选致病基因,在风险相关细胞类型(前脂肪细胞和脂肪细胞)前10个IR位点附近筛选了区域性和生物学上可行的16个基因。
Introduction
胰岛素抵抗(Insulin resistance, IR)在二型糖尿病(Type 2 diabetes mellitus, T2D)和心血管疾病(Cardiovascular disease, CVD)的病理生理中都起到关键作用。IR的临床危险因素包括肥胖,血脂异常,炎症,高胰岛素血症和血糖异常。除了这些经典的IR临床危险因素外,遗传变异还可以通过调节上述危险因素直接或间接地调节IR的危险。IR的遗传风险位点注释已对T2D和CVD的病因产生了见解,有助于心脏代谢疾病的预测,预防和个性化治疗。
全基因组关联研究(Genome-wide association studies, GWAS)已确定约60个与IR风险相关的位点。在两项GWAS研究中前10个重复的IR相关位点且与T2D相关。它们分别位于PPARG (the lead SNP, rs17036328)、IRS1 (rs2943645)、GRB14 (rs10195252)、PEPD (rs731839)、PDGFC (rs6822892)、MAP3K1 (rs459193)、ARL15 (rs4865796)、FAM13A (rs3822072)、RSPO3 (rs2745353) 和 LYPLAL1 (rs4846565) 的非编码区。多基因风险评分(IR-score)表明,皮下脂肪组织(尤其是脂肪酸)充当脂质能量平衡的缓冲系统,并在代谢和心血管疾病风险中起保护作用。
尽管GWAS在识别与IR相关的遗传位点方面取得了成功,但在每个位点中找出致病基因仍然是一个挑战。近年来,染色体构象捕获(Chromosome conformation capture, 3C)技术和表达数量性状位点(Expression quantitative trait loci, eQTL)的研究已经发现了GWAS位点与局部或远端基因之间的结构和功能联系。然而:
- 3C实验费用昂贵。
- eQTL研究不能识别出一个位点的所有效应基因,也不能准确定位疾病危险位点的致病基因和机制。
本研究采用的替代策略,通过研究风险相关细胞类型中候选基因的疾病相关功能,对疾病相关基因位点中的候选致病基因进行优先排序。为此,作者开发了一个体外敲除(KO)筛选平台,并对人前脂肪细胞和脂肪细胞中的16个IR候选基因进行了功能评估,以验证候选基因并发现其潜在的分子机制。在16个候选基因中,除PPARG和IRS1外,其余14个候选基因在人前脂肪细胞和脂肪细胞中功能尚未明确。
作者在人辛普森-戈拉比-贝梅尔综合征(Simpson-Golabi-Behmel syndrome, SGBS)前脂肪细胞中分别靶向候选基因。SGBS前脂肪细胞细胞株来源于SGBS患者的脂肪组织标本。它们提供了无限的成脂细胞来源以及基因编辑的机会,因为它们能够在保留成脂分化能力的同时增殖50代。SGBS-KO前脂肪细胞和SGBS-KO脂肪细胞被用于检测风险相关表型,包括脂肪生成、脂代谢和对胰岛素的敏感性,这可能是脂肪组织和其他组织类型,如肌肉、肝脏、心脏和胰腺的IR致病机制。作者使用增殖和分化实验研究了KO前脂肪细胞的脂肪生成,通过测量甘油三酯(TG)储存和脂解水平研究KO脂肪细胞的脂代谢,以及通过检查胰岛素诱导AKT2磷酸化和葡萄糖摄取研究KO脂肪细胞的胰岛素敏感性,旨在发现脂肪谱系的IR候选致病基因。这导致鉴定出12个候选基因,这些基因的敲除导致异常脂肪生成、脂质代谢和/或胰岛素信号。作者的研究结果强调了FST、PEPD和PDGFC在前脂肪细胞和脂肪细胞中调节IR的潜在新作用。这些结果表明,T2D和CVD的新的病因机制与这些尚未充分研究的基因功能有关。
Generation of SGBS preadipocyte knockout lines
作者选择了在前脂肪细胞和脂肪细胞都有表达的16个候选基因,其中除IRS1和PPARG(在本研究中作为阳性对照)外,人体脂肪组织中大多数IR基因的功能都有待研究。此外,使用了SLC2A4作为额外对照用于胰岛素诱导葡萄糖摄取。
总的来说,使用了17个基因(16个IR基因和SLC2A4),利用CRISPR/Cas9生成SGBS-KO前脂肪细胞细胞系以及一个使用杂乱sgRNA(SCR)的对照细胞系。
Figure 1. Examination of gene knockout efficiency at the genomic level in Simpson-Golabi-Behmel syndrome (SGBS)-preadipocytes.
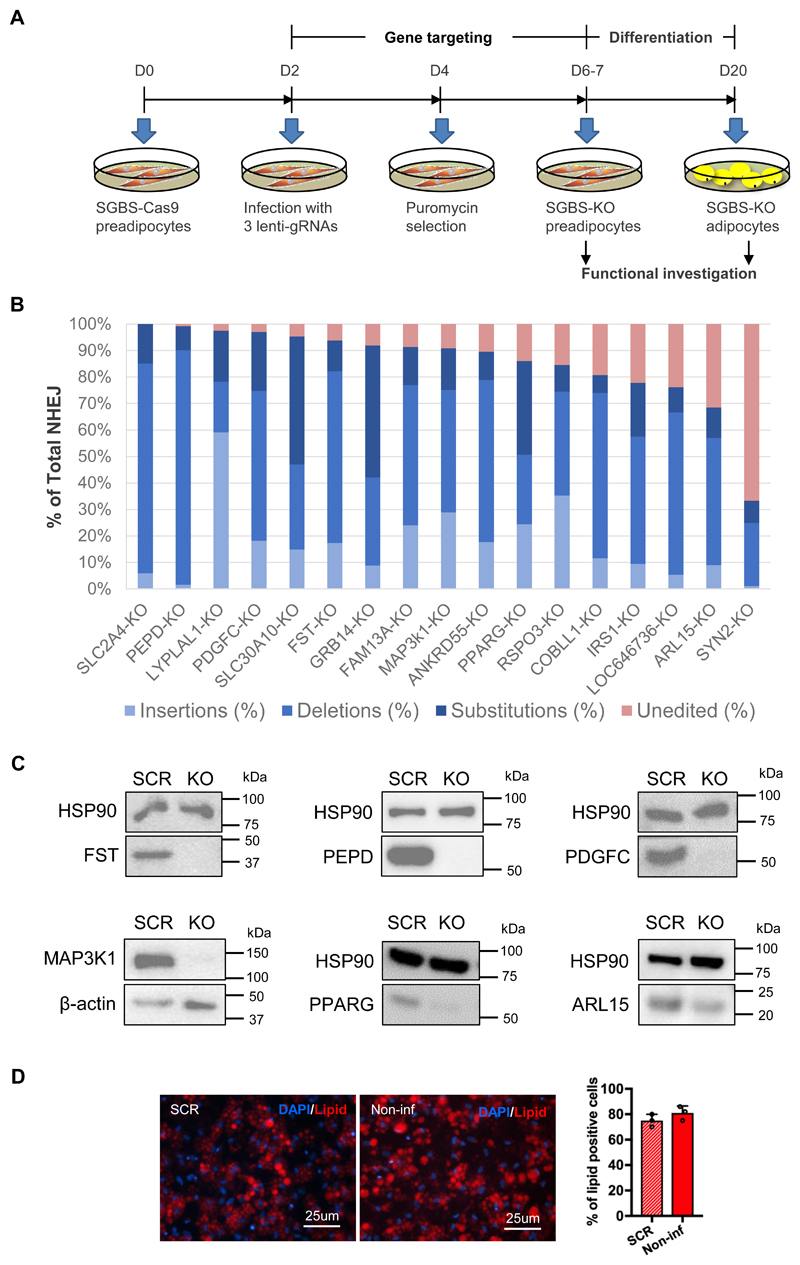
- (A)SGBS细胞的CRISPR/Cas9打靶、分化和功能研究的实验示意图。为了生成每个SGBS-KO细胞系,作者使用三种向导RNA方法靶向表达Cas9的SGBS细胞系中每个基因的一个或两个外显子。为了量化CRISPR/Cas9系统在敲除前脂肪细胞系中的靶向效率,作者扩增了靶基因位点并对扩增子进行测序。
- (B)使用二代测序(NGS)定量目标位点的敲除效率。在柱状图上,蓝色层次条表示三种类型的非同源末端连接(NHEJ),即插入、缺失和替换的总百分比。顶部红色条表示未编辑序列的百分比。
- (C)通过WB在蛋白水平确认基因敲除。如图显示了FST-、PEPD-、PDGFC-、MAP3K1-、PPARG-和ARL15-KO细胞系。图上的ARL15-KO脂肪细胞中,蛋白表达减少但未完全消除,与图1B的65%的编辑效率一致。
- (D)杂乱(SCR)和未感染(non-inf)SGBS-Cas9前脂肪细胞的分化效率。红色为脂类染液、蓝色为DAPI。柱状图显示了两种细胞系中脂类阳性细胞占脂肪细胞的百分比。CRISPR/Cas9可能导致DNA损伤应答,这种影响通常发生在第一代细胞。因此作者使用了第三代SGBS-KO前脂肪细胞,由图可知,慢病毒感染并未影响脂肪生成。
Roles of IR-genes in adipogenesis
前脂肪细胞的增殖和脂质储存脂肪细胞的形成是脂肪生成的两种主要步骤,因此也是IR风险相关的细胞功能。
因此作者:
- 测量了SGBS-KO前脂肪细胞的扩增能力。
- 研究了16种候选IR基因在SGBS-KO前脂肪细胞分化中的影响。
Figure 2. Knockout (KO) of insulin resistance-genes affects adipogenesis of preadipocytes.
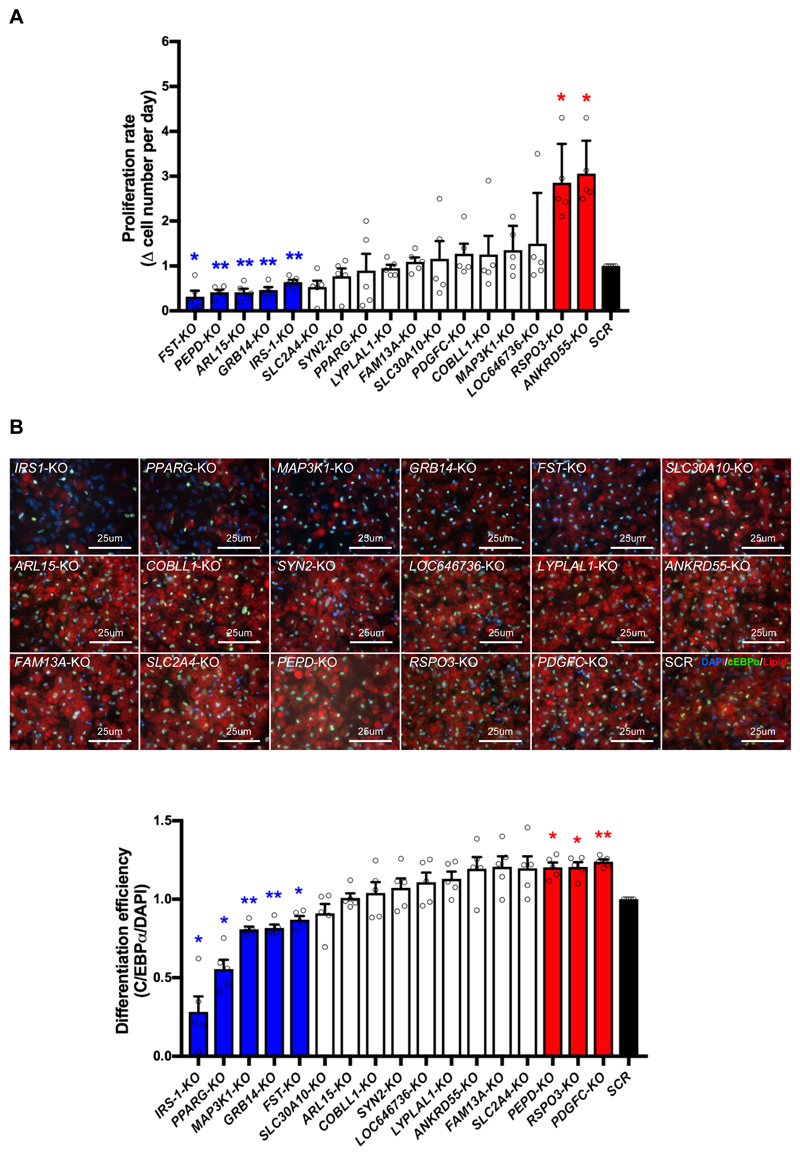
- (A)每种SGBS-KO前脂肪细胞系的增值率,通过每天的细胞数增加来计算。IRS1是促进脂肪祖细胞增殖的基因,如预期其KO细胞系增值率减少。
- (B)IR基因对SGBS前脂肪细胞分化影响的可视化研究。上图显示C/EBPα(绿色)、DAPI(蓝色)和脂滴(红色)染色的基因KO脂肪细胞的伪彩色图像。下图显示了每个SGBS-KO前脂肪细胞系的分化效率,计算结果为在DAPI阳性细胞中C/EBPα表达细胞的百分比。蓝色和红色条分别表示SGBS-KO细胞系分化效率降低与增高,黑色条表示SCR对照,白色条显示了与SCR对照没有显著差异的数据。如预期那样,PPARG-KO和IRS-1-KO分化效率降低,这与PPARG作为脂肪生成主调控因子的作用和IRS-1上调PPARG和C/EBPα表达的作用一致。
成脂转录因子CCAAT/增强子结合蛋白(CCAAT/enhancer binding protein α, C/EBPα)
通用的前脂肪细胞的分化标记物。
总的来说,IR基因的10个敲除系包括PPARG、IRS-1、GRB14、FST、PEPD、PDGFC、MAP3K1、ARL15、ANKRD55和RSPO3,显示出前脂肪细胞增殖和/或分化的缺陷。这些基因的异常表达可能导致脂肪生成过多或不足,并且两种极端情况都可能导致IR。但是,IR风险评分仅与皮下脂肪组织(SAT)质量减少有关,这表明IRS-1,PPARG,GRB14和FST的下调以及RSPO3,PDGFC和ANKRD55的上调可能有助于这种关系。
Roles of IR-genes in lipid metabolism
十个IR位点和相应的多基因风险评分与血脂异常相关,包括高甘油三酯和低高密度脂蛋白(HDL)水平,这也是IR的两个标志。血脂异常导致肝脏,骨骼肌和其他非SAT组织的异位脂质积累,这有助于IR在这些组织发生。为了研究IR基因在脂肪细胞脂代谢中的作用,作者测定了SGBS-KO脂肪细胞的脂质储存和脂肪分解。
Figure 3. Knockout (KO) of insulin resistance-genes affects lipid metabolism of adipocytes.
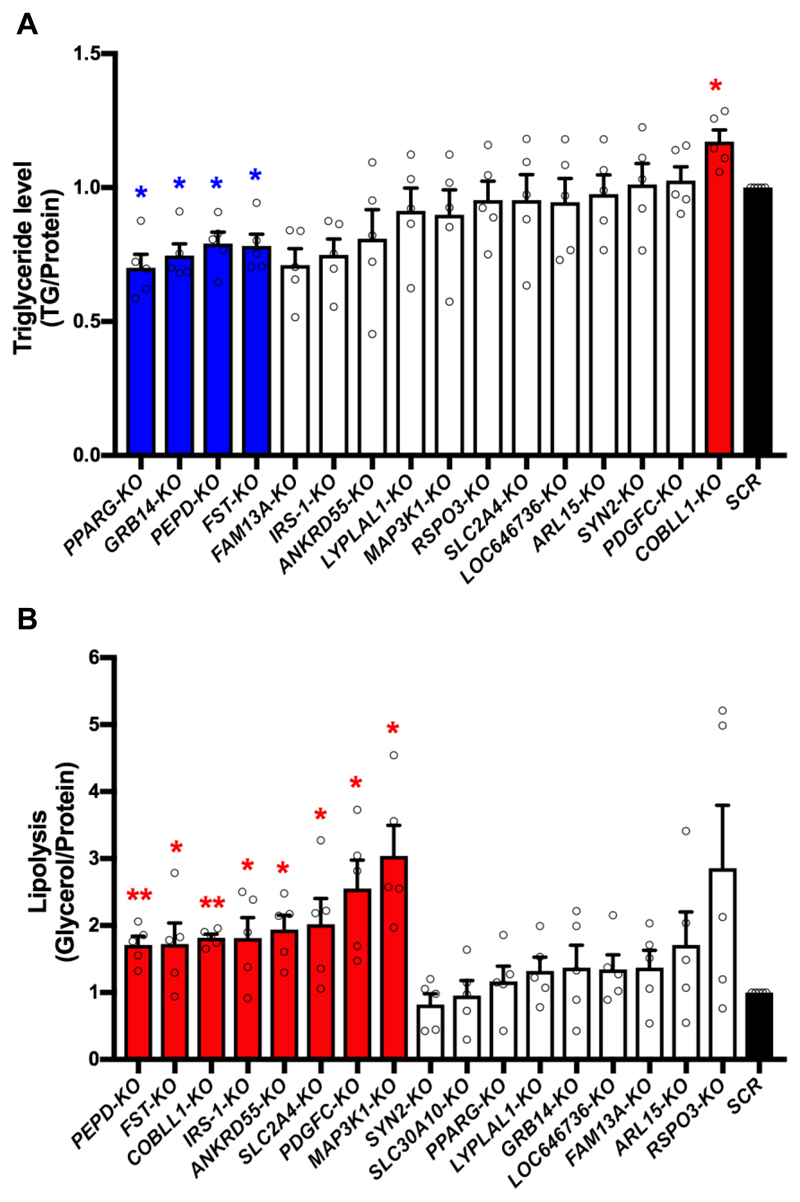
- (A)作者通过将细胞甘油三酯(TG)含量与总蛋白量进行标准化,评估了SGBS-KO脂肪细胞的脂质储存能力。正如预期的那样,PPARG-KO降低了SGBS-KO脂肪细胞的TG储存能力。敲除GRB14、PEPD和FST也降低了SGBS-KO脂肪细胞的TG积累。敲除COBLL1增加了TG的积累,表明COBLL1在脂质代谢中发挥了新的作用。
- (B)每个KO系的脂肪分解是通过甘油释放与总蛋白量归一化来评估的。KO-脂肪细胞的基础脂肪分解低于检测限。因此作者使用10μM的佛司可林(forskolin)来诱导脂肪分解。该实验显示,与SCR对照脂肪细胞相比,8个SGBS-KO细胞系的脂肪分解增加,包括PEPD、FST、COBLL1、IRS-1、ANKRD55、SLC2A4、PDGFC和MAP3K1的KO系。
除了增加脂肪分解,PEPD-和FST-KO还降低了TG的储存量,方向一致的表型表明,PEPD和FST表达的扰动很可能导致血脂异常,并可能与脂肪代谢障碍的多基因贡献有关。敲除COBLL1导致SGBS-KO脂肪细胞中的脂质积累和脂肪分解,表明COBLL1表达的减少可能导致类似肥胖的血脂异常。
脂肪分解是甘油三酯被水解成甘油和游离脂肪酸的代谢途径。
脂肪代谢障碍是一种由缺乏功能性脂肪组织引起的疾病,驱动着严重形式的IR。
Roles of IR-genes in insulin receptor signaling
10个IR位点的风险等位基因与较高的空腹胰岛素(fasting insulin, FI)水平相关。为了评估16个候选基因对胰岛素敏感性的影响,作者探究了IR基因KO对SGBS-KO脂肪细胞中胰岛素信号激活和胰岛素反应的葡萄糖摄取的影响。在脂肪细胞中,胰岛素激活胰岛素受体,启动涉及AKT2磷酸化的信号传导级联反应。AKT2是脂肪代谢的中央信号调节器,因此作为作者的候选基因验证方法的一部分,继而评估了胰岛素诱导的SGBS-KO脂肪细胞中的AKT2磷酸化。
为了进一步测试胰岛素信号传导的下游功能是否会受到基因敲除的影响,作者测量了胰岛素刺激后的细胞葡萄糖摄取量。胰岛素刺激的葡萄糖摄取是脂肪组织的主要生理功能,部分受AKT2 调节。循环葡萄糖以TGs的形式储存在脂肪细胞中,降低血糖水平,避免异位脂肪堆积。一旦葡萄糖被运输到细胞中,就会在糖酵解途径中被消耗掉。2-脱氧葡萄糖(2-DG)是葡萄糖的衍生物,可被己糖激酶磷酸化,形成2-脱氧葡萄糖-磷酸(2-DG-P)。2-DG-P不能在糖酵解的后续步骤中代谢,导致其在细胞中的积累,并提供了一个理想的替代品,用于测量胰岛素诱导的葡萄糖摄取。
Figure 4. Knockout (KO) of insulin resistance-genes perturbs the insulin sensitivity of adipocytes.
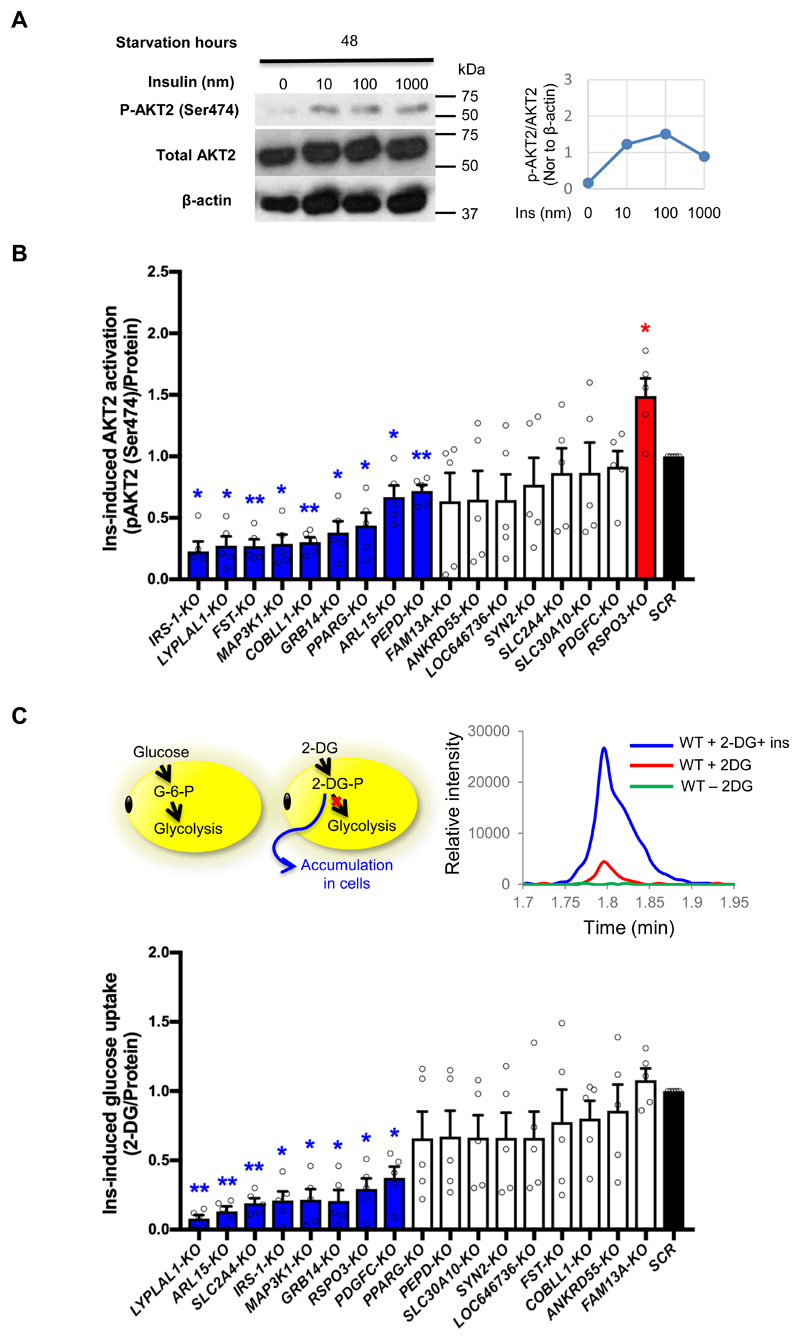
- (A)饥饿48小时后SGBS脂肪细胞的胰岛素反应。作者评估了0、10、100和1000 nm胰岛素(Ins)处理后SGBS脂肪细胞的磷酸化AKT2(Ser474)、总AKT2和β-肌动蛋白的水平。线图显示的是磷酸化AKT2(Ser474)与总AKT2的比值,并将该比值归一化为同一样品的β-肌动蛋白水平。两天的饥饿减弱了SGBS脂肪细胞中超刺激的p-AKT2,10 nm胰岛素处理后p-AKT2水平升高,100 nm胰岛素处理后达到饱和。
- (B)作者使用Elisa检测了胰岛素刺激的SGBS-KO脂肪细胞中AKT2的磷酸化情况。将Ins诱导的SGBS-KO脂肪细胞的磷酸化AKT2(Ser474)水平通过饥饿后10 nm胰岛素诱导的p-AKT2(Ser474)水平的变化来计算,结果归一化为总蛋白量,并以柱状图显示。发现与SCR对照组脂肪细胞相比,IRS-1、LYPLAL1、FST、MAP3K1、COBLL1、GRB14、PPARG、ARL15和PEPD的KO组AKT2磷酸化减弱,RSPO3的KO组增强了胰岛素诱导的AKT2磷酸化。
- (C)
- 左上角描述了脂肪细胞对两种类似物D-葡萄糖和2-脱氧葡萄糖(2-DG)的摄取。
- 右上图显示了WT SGBS-脂肪细胞在三种条件下对2DG-6-P的摄取质谱图,通过量化细胞内2-DG-P,并比较有和没有胰岛素处理的细胞之间的摄取。胰岛素处理WT SGBS脂肪细胞30分钟后,与未处理的对照组相比,细胞的2-DG-P水平增加了5-6倍。
- 底部图显示胰岛素诱导的SGBS-KO脂肪细胞的葡萄糖摄取量,作为10 nm胰岛素处理30min后的2-DG-6-P水平,归一化为总蛋白量。8个IR基因敲除(LYPLAL1、ARL15、SLC2A4、IRS1、MAP3K1、GRB14、RSPO3和PDGFC)降低了SBGS-KO脂肪细胞对胰岛素刺激的葡萄糖摄取。SLC2A4-KO也降低了SGBS-KO脂肪细胞的葡萄糖摄取,这是因为缺乏胰岛素调节的葡萄糖转运体GLUT4。IRS-1、MAP3K1、GRB14、LYPLAL1和ARL15同时促进了胰岛素反应性AKT2磷酸化和葡萄糖摄取,而PDGFC-KO降低了胰岛素刺激的葡萄糖摄取,但不影响AKT2磷酸化。
Mechanistic evaluation of candidate causal genes
通过体外筛选,作者获得了7个候选基因。
随后通过GTEx (V7) eQTL计算器进行分析,确定了6个eQTL基因。可以看到,与体外筛选的基因有5个是相对应的,包括IRS-1、GRB14、PDGFC、PEPD和FST。可以看到IRS1、FST、PEPD和PDGFC的表达是下调的,剩下的为上调。
随后作者利用T2D Knowledge Portal中存储的数据分析了6个顺式eQTL的风险等位基因对IR、T2D和CVD的影响,发现它们与较高的FI和T2D风险相关,有与CVD风险增加相关的趋势。
Figure 5. Overlap of genes acting on the 6 cell functions tested in preadipocytes or adipocytes and genetic analysis of expression quantitative trait loci (eQTL) genes.
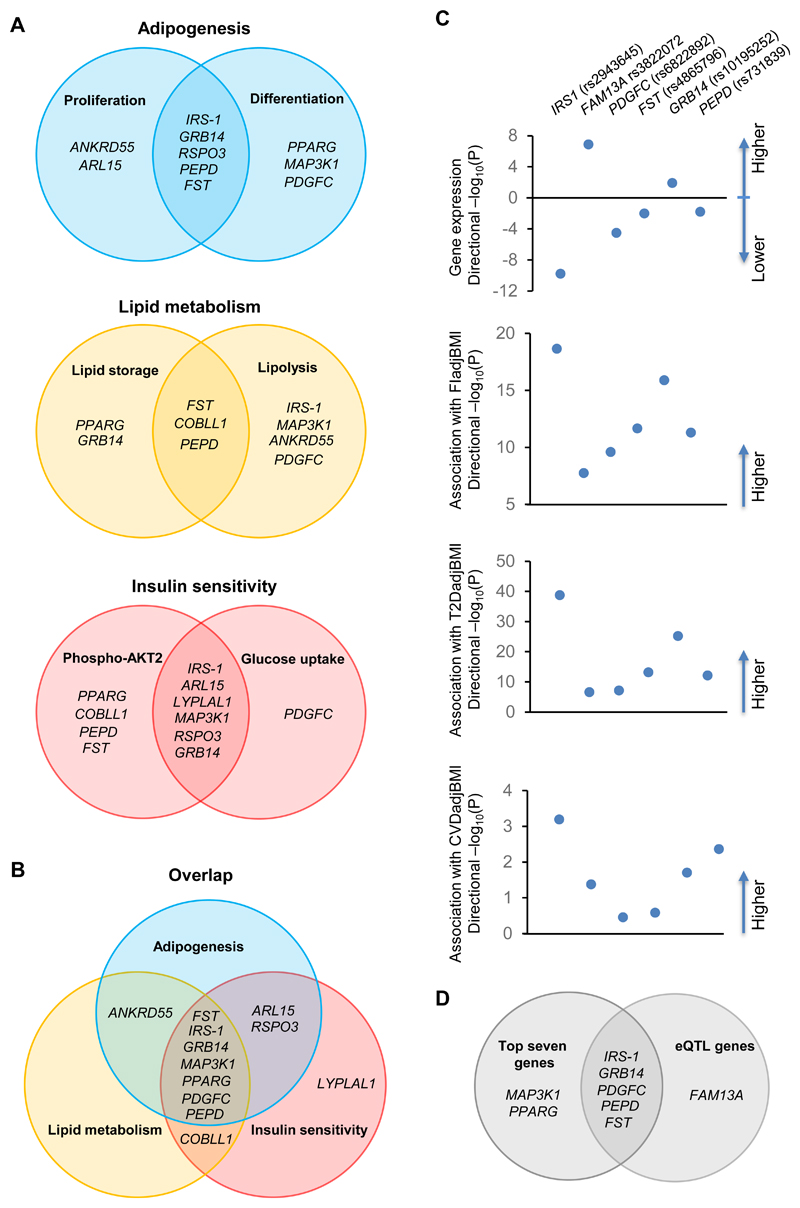
- (A-B)Venn图说明了在六种IR风险相关细胞功能中表现出表型的IR基因。FST、IRS-1、GRB14、MAP3K1、PPARG、PDGFC和PEPD这7个基因在这3个机制中都表现出功能,将它们评为IR的首要候选致病基因。
- (C)在每个IR位点的主导SNP的风险等位基因与eQTL基因的表达的关联:
- (I)GTEx数据库V7的人类皮下脂肪组织;
- (II)空腹胰岛素(FI);
- (III)T2D;
- (IV)CVD。
- (D)从eQTL分析的基因和体外筛选的前7个基因之间的重叠候选基因。找到了5个基因:IRS-1、GRB14、FST、PEPD和PDGFC。
GTEx(Genotype-Tissue Expression)
该项目对来自人体多个组合和器官的样本,同时进行了转录组测序和基因分型分析,构建了一个组织特异性的基因表达和调控的数据库。
eQTL
eQTL主要是获得在特定组织中单个SNP和单个基因表达的相关性分析,即获得的是单个基因mRNA表达量。eQTL可分为顺式作用eQTL和反式作用eQTL,顺式作用eQTL就是某个基因的eQTL定位到该基因所在的基因组区域,表明可能是该基因本身的差别引起的mRNA水平变化;反式作用eQTL是指某个基因的eQTL定位到其他基因组区域,表明其他基因的差别控制该基因mRNA水平的差异。
Figure 6. Phenotypes of gene-knockout (KO) preadipocytes and adipocytes.
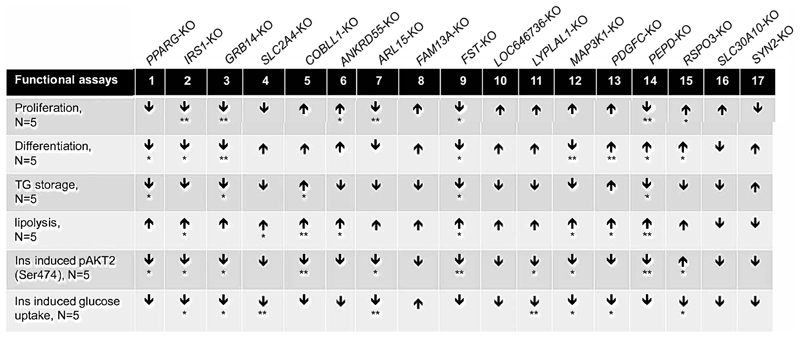
基因-KO前脂肪细胞和脂肪细胞的表型。
Phenotypic rescue in FST-, PEPD- and PDGFC-KO preadipocytes and adipocytes
在这项研究中发现的5个功能基因和eQTL基因中,FST、PEPD和PDGFC以前与脂肪胰岛素敏感性无关。为了进一步证实FST、PEPD和PDGFC基因敲除导致的表型,作者研究了CRISPR-Cas9基因组编辑策略的特异性,并拯救了这三个基因在相应KO系中的表达。
作者在相应的基因KO细胞中进行FST、PEPD和PDGFC的表达拯救。将这三个基因的开放阅读框(ORF)克隆到商业mRNA表达质粒中。通过Western Blot证实了FST、PEPD和PDGFC在相应的KO-脂肪细胞中的转基因表达,尽管mRNA转染没有将蛋白水平重建到与SCR细胞系一致的水平。
拯救实验被设计为专门探究各自SGBS-KO细胞系的受影响功能。作者进行了增殖、分化、TG储存、脂肪分解和胰岛素反应性AKT2磷酸化的测定,以评估FST-KO和PEPD-KO细胞系的表型拯救。
Figure 7. Phenotypic rescue in knockout (KO) lines of FST (follistatin; A), PEPD (Xaa-Pro dipeptidase or prolidase; B), and PDGFC (platelet-derived growth factor C; C).
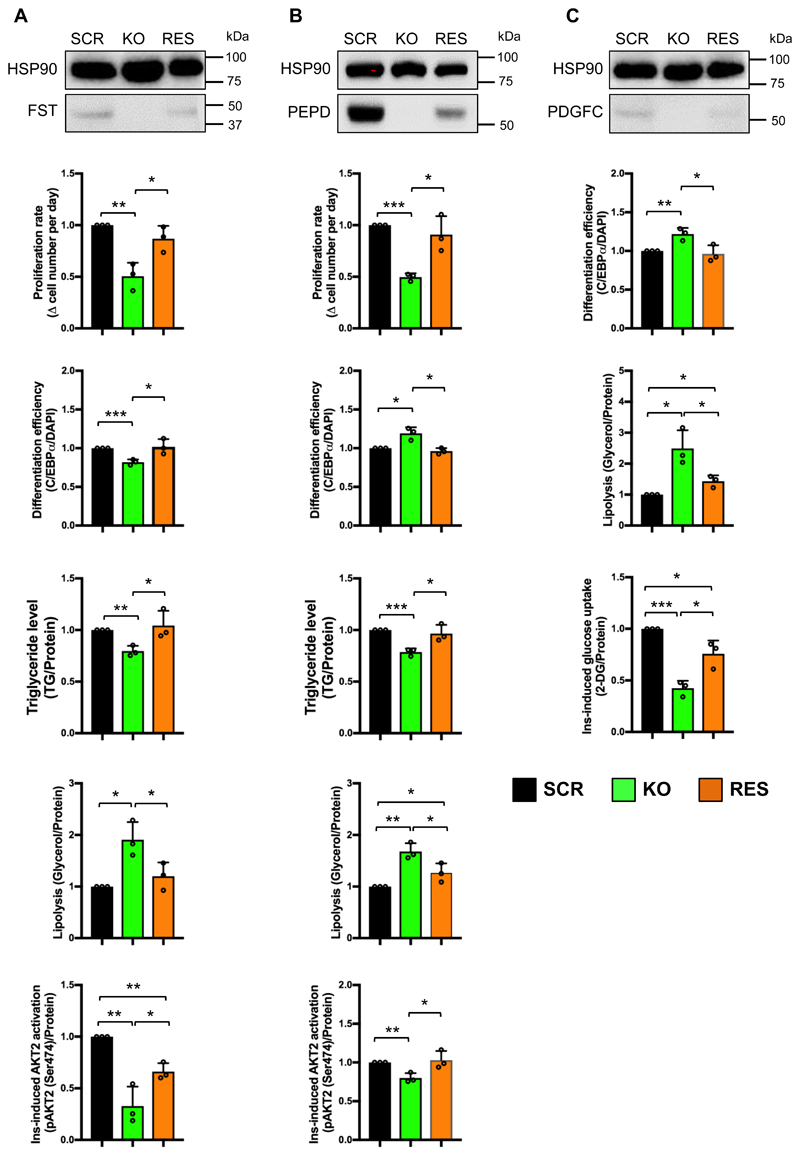
mRNA介导的拯救显著改善了三个KO细胞系的扰动表型,与转基因表达水平一致。结果表明,FST-、PEPD-和PDGFC-KO前脂肪细胞和脂肪细胞的功能扰动是由于CRISPR/Cas9对这三个基因进行了靶向基因组编辑,而不是由于任何脱靶事件。其他KO细胞系的sgRNA设计策略完全相同,说明在这些细胞系中观察到的表型也是靶向基因组编辑的潜在结果。这些表型拯救实验进一步强调了FST、PEPD和PDGFC在前脂肪细胞和脂肪细胞的脂肪发生、脂质代谢和胰岛素信号传导中的意义。
Discussion
自2005年GWAS时代开始以来,数以千计的位点在统计上与多基因疾病和性状的风险相关。而相关下游的生物学研究仍有待探索,例如虽然约60位点与IR的风险相关,但大多数位点仍未研究。
T2D和CVD有许多共同的风险因素,但很少有风险位点(约10个)在两种疾病之间共享,具有基因组广泛的意义。然而,IR风险等位基因IRS1、PEPD、GRB14、FAM13A、FST和PDGFC,均与T2D风险相关,并在较小程度上与CVD风险相关,提示IR在T2D和CVD之间链接了共同的病因学途径。结合筛选结果和网络分析的eQTL以及GWAS研究,强调了前7个候选基因(IRS-1、PPARG、GRB14、PEPD、FST、PDGFC和MAP3K1)和FAM13A在脂肪发生或脂肪组织扩张中的功能,这可能是候选致病基因与IR、T2D和CVD风险之间关联的桥梁。这些结果通过提供IR基因与前脂肪细胞和脂肪细胞功能之间的机制联系,增强了脂肪组织在心脏代谢疾病中功能障碍的现有证据。
作者的功能研究重述了IRS和PPARG在脂肪发生、脂质代谢和胰岛素信号传导中的关键作用,这些已知机制与IR、T2D和CVD的发病机制有关。在这两个GWAS位点的许多遗传变异与脂肪发育相关的心脏代谢表型的风险相关,表明结合遗传分析和相关功能研究可能是一个可靠的方式来发现新的基因和机制控制疾病风险。在这项研究中,作者发现了前脂肪细胞和脂肪细胞中候选基因的新功能。
MAP3K1,又称MEKK1,是一种丝氨酸和苏氨酸激酶。GWAS和细粒度图谱确定MAP3K1为乳腺癌的致病基因。MAP3K1 KO-脂肪细胞增殖率和脂肪分解增加,这与皮下脂肪组织中MAP3K1的下调和儿童肥胖和T2D关联的事实一致。这里确定的候选致病IR基因可能为心脏代谢疾病的新机制提供启示。
FST或follistatin直接与激活素A结合,起到激活素拮抗剂的作用。人类脂肪组织表达和分泌follistatin和激活素A,激活素A介导的信号转导促进细胞增殖,抑制脂肪生成,而follistatin抑制激活素A,促进脂肪生成。筛选结果和遗传分析都支持follistatin在脂肪发生中的重要性。此外,作者还发现,follistatin还可以在脂质代谢和胰岛素信号传导中发挥作用。
PEPD是一种a型脯氨酸酶,通过回收脯氨酸合成胶原蛋白和其他含脯氨酸的蛋白质,参与细胞外基质的形成和维持。一般来说,脯氨酸酶直接结合并激活表皮生长因子受体,刺激细胞生长和增殖。血清脯氨酸酶活性与冠状动脉疾病的存在和严重程度有关。作者发现PEPD可以促进前脂肪细胞的增殖和脂肪细胞中AKT2的磷酸化,同时抑制脂肪分解。该酶的原理功能表明,其在脂肪生成、脂质代谢和胰岛素信号传导中的作用可能是继发于其对细胞外基质的生物合成的作用,这是一种新提出的心脏代谢疾病的机制。
PDGFC是PDGF家族的成员之一,PDGF受体(PDGFRs)在血管系统中广泛表达,对血管的正常发育和维护非常重要。PDGFs通过PDGFRs的旁分泌信号作用。PDGFCs在脂肪组织中的表达可能涉及脂肪组织和局部血管系统之间的交叉对话,为组织发育和生长所需的血管生成。然而,由于在我们的体外系统中缺乏血管成分,PDGFC-KO细胞在脂肪生成分化,溶脂和胰岛素诱导的葡萄糖摄取的缺陷可能表明未知的功能。事实上,PDGFRs也表达在前脂肪细胞和脂肪细胞。另一方面,GWAS和皮下脂肪组织的eQTL研究都表明PDGFC在胰岛素敏感性和T2D中具有保护作用。这些数据可以指出PDGFC和血管生成在脂肪扩张中的作用, 因此在IR和心脏代谢表型中也起作用 。
综上所述,功能筛选优先确定了12个基因作为前脂肪细胞和/或脂肪细胞IR的候选致病基因。作者的体外和遗传数据共同强调了PPARG、IRS-1、GRB14、FST、PEPD、PDGFC、MAP3K1和FAM13A对IR的致病作用。目前的机理研究提示了它们在前脂肪细胞和脂肪细胞中的作用,这可能是它们与疾病风险关联的桥梁。目前对脂肪组织中FST、PEPD和PDGFC的功能了解甚少,这可能涉及心脏代谢疾病的新机制。
Reference
Chen Z F, Yu H J, Shi X, et al. Functional Screening of Candidate Causal Genes for Insulin Resistance in Human Preadipocytes and Adipocytes[J]. Circ Res, 2020, 126(3): 330-346.